10x 空间转录组数据跑完 Space Ranger 之后都会生成单个样本的 cloupe.cloupe 文件,这个文件可以用 Loupe Browser 来对单个样本进行可视化,Loupe Browser 操作简单、结果直观,非常适合不太会写代码的老师或同学来自己挖掘数据。但是,做过单细胞或空间转录组项目的同学应该都知道,实际上做数据分析的时候是需要多个样本合并起来分析的,如果再用单个样本进行可视化就不太合适了,所以我们需要把多个样本的 cloupe.cloupe 整合成一个合并的 cloupe 文件,这样就方便自己用 Loupe Browser 来有效的挖掘有价值的信息。
这里我们来介绍一下,怎么用 10x spaceranger aggr 来合并多个空间转录组测序的样本数据。
步骤一
确保单个样本的 spaceranger count 已经跑完了,假如我们前面运行 spaceranger count 时生成了三个文件:
1 $ spaceranger count --id=LV123 ……
2 wait for pipeline to finish ……
3 $ spaceranger count --id=LB456 ……
4 wait for pipeline to finish ……
5 $ spaceranger count --id=LP789 ……
6 wait for pipeline to finish ……
步骤二
准备样本信息的 csv 文件,内容格式如下(注意逗号分隔)。
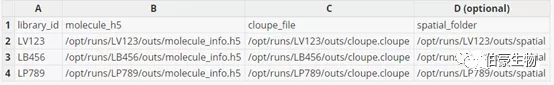
列信息说明:
library_id: 样本 id;
molecule_h5: 运行 spaceranger count 生成的 molecule_info.h5 文件路径;
cloupe_file: 运行 spacerangercount 生成的 cloupe.cloupe 文件路径;
spatial_folder: 运行 spaceranger count 生成的 spatial 文件夹路径。
除了上面指定的 4 列之外,也可以加入其它分类信息的列,之后会在 Loupe Browser 可视化的时候当成一个分组方式来展示。比如下面,增加一列样本分组信息,不过注意这里因为宽度的原因把 spatial_folder 列省略掉了,实际上 spatial_folder 这一列还是有的。
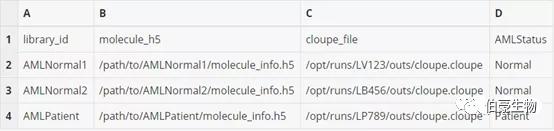
另外需要注意,spaceranger aggr 是不进行批次校准的,所以不同的染色方法的组织是不能合并到一起的(例如免疫荧光和 H & E 染色的组织切片)。
步骤三
运行 spaceranger aggr:
1 $ spaceranger aggr --id=AGG123 \
2 -- csv=AGG123_libraries.csv \
3 -- normalize=mapped
参数说明:
--id: 输出文件的 id
--csv: 样本信息 csv 文件
--normalize:depth 归一化的方法,默认是 mapped,也可以选 none。
结果输出:
成功运行后会到的下面的信息(包括主要的输出文件)
1 2018-10-04 13:36:33[runtime] (run:local)
ID.AGG123.SPATIAL_RNA_AGGREGATOR_CS.SPATIAL_RNA_AGGREGATOR.SUMMARIZE_AGGREGATED_REPORTS.fork0.join
2 2018-10-04 13:36:36 [runtime] (join_complete)
ID.AGG123.SPATIAL_RNA_AGGREGATOR_CS.SPATIAL_RNA_AGGREGATOR.SUMMARIZE_AGGREGATED_REPORTS
3 2018-10-04 13:36:45 [runtime] VDR killed 210files, 29MB.
4
5 Outputs:
6 - Aggregation metrics summary HTML: /home/jdoe/runs/AGG123/outs/web_summary.html
7 - Aggregation metrics summary JSON: /home/jdoe/runs/AGG123/outs/summary.json
8 - Secondary analysis output CSV: /home/jdoe/runs/AGG123/outs/analysis
9 - Filtered feature-barcode matrices MEX: /home/jdoe/runs/AGG123/outs/filtered_feature_bc_matrix
10 - Filtered feature-barcode matrices HDF5: /home/jdoe/runs/AGG123/outs/filtered_feature_bc_matrix.h5
11 - Unfiltered feature-barcode matrices MEX: /home/jdoe/runs/AGG123/outs/raw_feature_bc_matrix
12 - Unfiltered feature-barcode matrices HDF5: /home/jdoe/runs/AGG123/outs/raw_feature_bc_matrix.h5
13 - Copy of the input aggregation CSV: /home/jdoe/runs/AGG123/outs/aggregation.csv
14 - Loupe Browser file: /home/jdoe/runs/AGG123/outs/cloupe.cloupe
15 - Aggregated tissue positions list: /home/jdoe/runs/AGG123/outs/aggr_tissue_positions_list.csv
16 - Spatial folder containing spatial images and scalefactors: /home/jdoe/runs/AGG123/outs/spatial
17
18 Pipestance completed successfully!
伯豪生物 空间转录组测序 服务优势
高质量切片:切片、贴片经验丰富,针对不同组织优化了解决方案。
流程化分析:完善的分析流程,准确快速解析空间转录组数据。
标准化内控:丰富的实操经验构建了标准化的内控体系。
专业化团队:资深的技术团队具有多年项目方案设计、实验操作、售后分析等经验。
全流程服务:提供组织冷冻包埋、贴片、切片、透化、建库测序及数据分析的全套服务。
更多伯豪生物人工服务:


