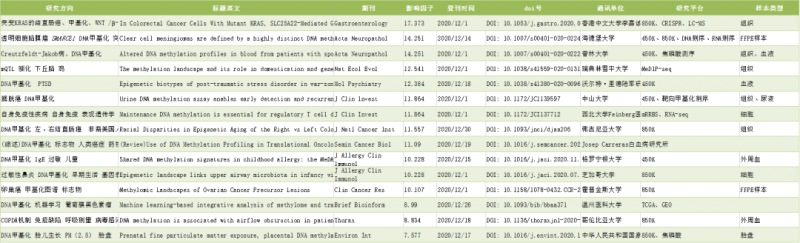
文章列表
虽然 2020 年已经过去一个月了,但是小编依然要为大家搬上 2020 年 12 月的 DNA 甲基化研究成果进行分享~
12 月以 DNA methylation 为关键词检索到的文献一共有 623 篇,小编挑出以下 6 篇作为重点分享内容。这 6 篇文章主要通过 450K、850K 技术从 DNA 甲基化层面揭示疾病发生的分子机制以及早筛标志物的发现(结直肠癌、透明细胞脑膜瘤、创伤后应激障碍、膀胱癌和卵巢癌),对后续想要做甲基化方向研究的老师,具有很好的参考价值。小编在文末附表中也列出更多 DNA 甲基化研究的文献和高分综述的研究方向、登刊时间、技术平台和摘要等信息,让您不用再费时费力搜索,就能轻松获取 12 月新的 DNA 甲基化研究成果和动态,平均 IF>10。

标题:在具有突变 KRAS 的结直肠癌细胞中,SLC25A22 介导的谷氨酰胺分解通过减少 DNA 脱甲基作用,从而增加 WNT 信号传导,干细胞特性和耐药性
时间:2020/12/01
单位:香港中文大学李嘉诚健康科学研究所
期刊:Gastroenterology 17.373
平台:850K、CRISPR、LC-MS
摘要:KRAS 突变会通过调控三羧酸循环途径促进谷氨酰胺分解,通过谷氨酰胺酶和 SLC25A22 将谷氨酰胺转化为 α - 酮戊二酸和其他分子。该过程会抑制细胞中脱甲基酶功能和改变表观遗传状态,从而增强细胞的增殖能力和干细胞特征。该研究主要是为了了解 KRAS 突变介导的谷氨酰胺分解是否影响大肠癌(CRC)细胞的表观基因组状态和活性。研究人员通过收集 SLC25A22 肠道特异性敲除小鼠——ApcminKrasG12D(ApcminKrasG12DSLC25A22fl/fl mice)的肠道组织,经组织学、免疫组化和 DNA 甲基化水平进行分析,并进一步研究来自 2 个人类结直肠肿瘤标本的类器官的来源和其干细胞特征。在 KRAS 突变体背景下的结肠上皮细胞(1CT)以及 CRC 细胞(DLD1,DKS8,HKE3 和 HCT116)中,表达或敲除 SLC25A22 或其他蛋白,并在没有谷氨酰胺或葡萄糖的条件下,进行增殖,群落形成,测定葡萄糖或谷氨酰胺消耗和细胞凋亡等表型的测定;通过 RNA 测序、免疫印迹、LC-MS(谷氨酰胺示踪)分别测定基因、蛋白质和代谢产物。同时,研究人员通过使用 850K 芯片分别测定敲除或过表达相关蛋白的细胞和类器官 CpG 位点的 DNA 甲基化水平。该团队检测了香港 130 例患者(57 例具有 KRAS 突变)的大肠癌样本的免疫组化和 Kaplan-Meier(生存期),并对 TCGA 数据库中大肠癌样本的相关基因表达水平进行分析。结果发现,在 KRAS 表达激活的 CRC 细胞中,SLC25A22 能够促进琥珀酸的积累,DNA 甲基化水平升高,WNT 信号向 β -catenin 的激活,LGR5 的表达增加,细胞增殖和干细胞特征以及对 5 - 氟尿嘧啶的抗性。通过阻断这一通路可能会被用于 CRC 的治疗。

标题:透明细胞脑膜瘤可以通过 SMARCE1 基因的 DNA 甲基化水平与突变类型成功分型时间:2020/12/14
单位:海德堡大学
期刊:Acta Neuropathol 14.251
平台:450K、850K、DNA 测序、RNA 测序
背景:透明细胞脑膜瘤是一种罕见的脑膜瘤变体,通常会影响儿童和年轻人。尽管已有报道称该亚型的 SMARCE1 基因中存在大量的功能缺失突变,但还缺乏全面的分子研究来证实。研究人员通过 WGBS 筛选了 3093 个脑膜瘤队列,其中大多数在组织学上被诊断为透明细胞脑膜瘤,并确定一个肿瘤亚群(n = 31)。该队列进一步补充了另外 11 种经组织学诊断出的透明细胞脑膜瘤进行分析(n = 42)。靶向 DNA 测序揭示了 33/34 个分析样品中存在 SMARCE1 突变,通过免疫组织化学确定的核表达缺失和肿瘤细胞中 SMARCE1 转录表达的降低。对透明细胞脑膜瘤组(n = 14)患者与 WHO 2 级脑膜瘤(n = 220)患者的进展或复发时间进行分析,两者结果相似。作者的发现表明透明质膜脑膜瘤存在高度不同的表观遗传学特征,与其他所有脑膜瘤变体不同的是 SMARCE1 基因具有反复突变。表明这些肿瘤可能源自与其他脑膜瘤亚型的不同的前体细胞群。

标题:退伍军人和现役男性中创伤后应激障碍的表观遗传型
时间:2020/12/18
单位:沃尔特·里德陆军研究所
期刊:Mol Psychiatry 12.384
平台:450K
背景:创伤后应激障碍(PTSD)是一种异质性疾病,目前缺乏适用于所有符合该疾病标准人的客观生理指标以及对治疗的不同反应。这项研究发现集纳入了 83 名患有 PTSD 的男性战斗退伍军人和 83 名没有 PTSD 的战斗退伍军人,利用 450K 检测其甲基化水平。DNA 甲基化(DNAm)分布图的计算分析确定了 PTSD + 组中的两种 PTSD 亚型——G1 和 G2,这两种亚型与 PTSD 和 PTSD 合并症相关的 34 种临床特征有关。G2 亚型与 G1 亚型和健康对照组相比,G2 亚型与 PTSD 风险增加相关,并且多基因风险评分更高且甲基化水平增加。接着,研究人员对同一个体进行了为期 3 年的随访(N = 59)并在两个独立的资深队列(N = 54 和 N = 38)和现役队列(N = 133)中进行进一步验证。在某些情况下,例如多巴胺 -PKA-CREB 和 GABA-PKC-CREB 信号通路,G2 亚型中两条通路的失调情况相反,表明该亚型的功能不只是与症状严重程度相关,也可能代表不同的生物风险轮廓支撑 PTSD。鉴别两种新的不同的 PTSD 表观遗传生物类型可能对了解 PTSD 的生物学和临床异质性、在非临床医生管理的环境下现役军人的风险评估和改进 PTSD 诊断标志物具有潜在的应用价值。

标题:尿液 DNA 甲基化测定可对膀胱癌进行早期检测和复发监测
时间:2020/12
单位:中山大学
期刊:J Clin Invest 11.864
平台:450K、靶向甲基化测序
背景:当前用于检测和监视膀胱癌(BCa)的方法通常是侵入性的或具有次优的敏感性和特异性,尤其是在早期肿瘤较小和残留肿瘤中。作者开发了一种有效的方法,称为 utMeMA,用于检测膀胱癌 MassARRAY 在多个基因组区域对尿液肿瘤 DNA 甲基化。作者通过对中山纪念医院(SYSMH),癌症基因组图谱(TCGA)和基因表达综合(GEO)数据库的队列进行综合分析,确定了 BCa 特异性甲基化标记。在回顾性队列中(n = 313)建立 BCa 诊断模型,并在多中心,前瞻性队列(n = 175)中进行了验证。分析了该模型诊断测定的性能,并将其与尿液细胞学和 FISH 进行了比较。结果发现,作者在联合分析中找到了 BCa 的 26 个重要的甲基化标记。并用其中的 2 个标记建立诊断模型并进一步验证,该模型能够以较高的准确度(86.7%),敏感性(90.0%)和特异性(83.1%)区分 BCA 患者。此外,基于 utMeMA 的检测相对于尿液细胞学和 FISH 的灵敏度有了很大提高。该研究提供的尿液中的肿瘤 DNA 甲基化评估可快速、高通量、无创地诊断尿液中的肿瘤 DNA 甲基化,从而对 BCa 进行早期诊断,检测和监测小限度的残留肿瘤。

标题:右 vs 左结肠的表观遗传老化中的种族差异
时间:2020/12/30
单位:弗吉尼亚大学
期刊:J Natl Cancer Inst 11.557
平台:850K
摘要:结直肠癌的发病年龄和偏侧性存在种族差异。假定表观遗传的年龄加速是一个潜在因素,然而,缺乏对侧面特异性结肠组织表观遗传衰老的比较研究。在这里,作者对来自 128 个个体的正常结肠左,右活检组织进行了 DNA 甲基化分析。在非裔美国人(n = 88)中,与个体匹配的左结肠相比,右结肠显示出加速的表观遗传衰老。相反,在欧洲裔美国人(n = 40)中,右结肠显示出明显的年龄减慢。进一步研究表明,DNA 甲基化的表观基因组范围分析确定了非裔美国人右结肠超甲基化的独特模式。作者的研究初次报道了正常结肠表观遗传衰老的种族和侧方特异性差异,为观察到的更年轻的发病年龄和非裔美国人右侧结肠瘤变的相对优势提供了新的见解。

标题:卵巢癌前体病变的甲基化景观
时间:2020/12
单位:霍普金斯大学
期刊:Clin Cancer Res 10.107
平台:850K
摘要:目前在发展高级别浆液性卵巢癌(HGSC)的范例中,大多数 HGSC 源自输卵管的前浆液性输卵管上皮内癌(STIC)病变。这篇文章作者使用 850K 比较了 12 个 STIC 与配对的正常上皮组织,并利用无监督的层次聚类和多维分析对数据进行分析。结果鉴定出高可信度 STIC 特异性差异高甲基化区域,并将其与来自 23 个 HGSC 肿瘤和 11 个健康输卵管粘膜的 850K 数据进行了比较。无监督的分析表明,STICs 在很大程度上与 HGSCs 聚集在一起,但与正常的输卵管上皮明显不同。42 个甲基化区域显示出高可信度的 STIC 特异性,其中有 17 个甲基化区域可以与 HGSC 特异性差异甲基化区域重合,且在交集中的甲基化位点能够将 STIC 和 HGSC 样品与正常样品完全区分开。该项研究表明,大多数 STIC 在表观遗传上与 HGSC 相似,并且共有差异性高甲基化区域,因此有必要进一步评估其作为早期检测卵巢 HGSC 的生物标记物的潜力。
文章列表
文末列出了小编整理出的完整文献列表,如果您对其中一些文献更感兴趣,就请联系我们,获得完整的文章吧!(伯豪生物微信获取:17702139967)
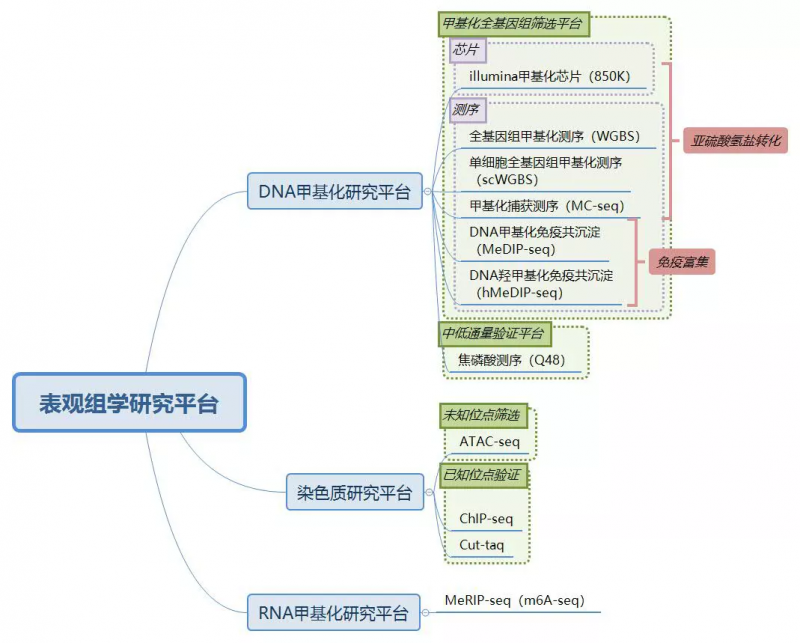
伯豪甲基化研究平台:
更多伯豪生物人工服务:


