差异表达(differentially expressed,DE)基因的检测和评估一直是单细胞 scRNA-seq 数据分析中关键的一步,准确可靠的 DE 基因结果对下游分析具有深远影响。scCODE 算法提出全新的单细胞 scRNA-seq 数据一站式个性化 DE 基因分析系统,通过对单细胞数据多次建模分析,自动评估优化分析结果,为科研人员深入解析单细胞数据提供准确高效的新工具。
2022 年 5 月 23 日, 复旦大学郝洁 / 邹欣团队与辰山植物园陈可 组在 Briefings in Bioinformatics(计算生物学一区,IF=11.6)上发表了题为:”scCODE: an R package for data-specific differentially expressed gene detection on single-cell RNA-sequencing data”的研究论文。该研究评估 40 余种 DE 分析策略,基于自主研发的,无需先验信息的两个新的评估参数,开发了 scRNA-seq 数据个性化 DE 基因筛选系统平台——scCODE(图 1)。

图 1:Evaluation of DE gene detection performance and schematic of scCODE.
在以往的研究中,过滤低表达的基因被认为有助于 DE 分析,但其真正影响尚未得知,基因过滤也无统一标准。本研究第一部分从 FPR、TPR 和 AUROC 等评估标准分析了不同基因过滤方法对 DE 结果的影响。结果发现,恰当的基因过滤和 DE 方法的组合可以得到更好的 DE 结果,而不恰当的组合却会起到相反的效果。当 DE 分析策略所推测的 DE 基因表达分布与实际的基因表达分布更接近时,才能得到更准确的 DE 基因结果(图 2)。
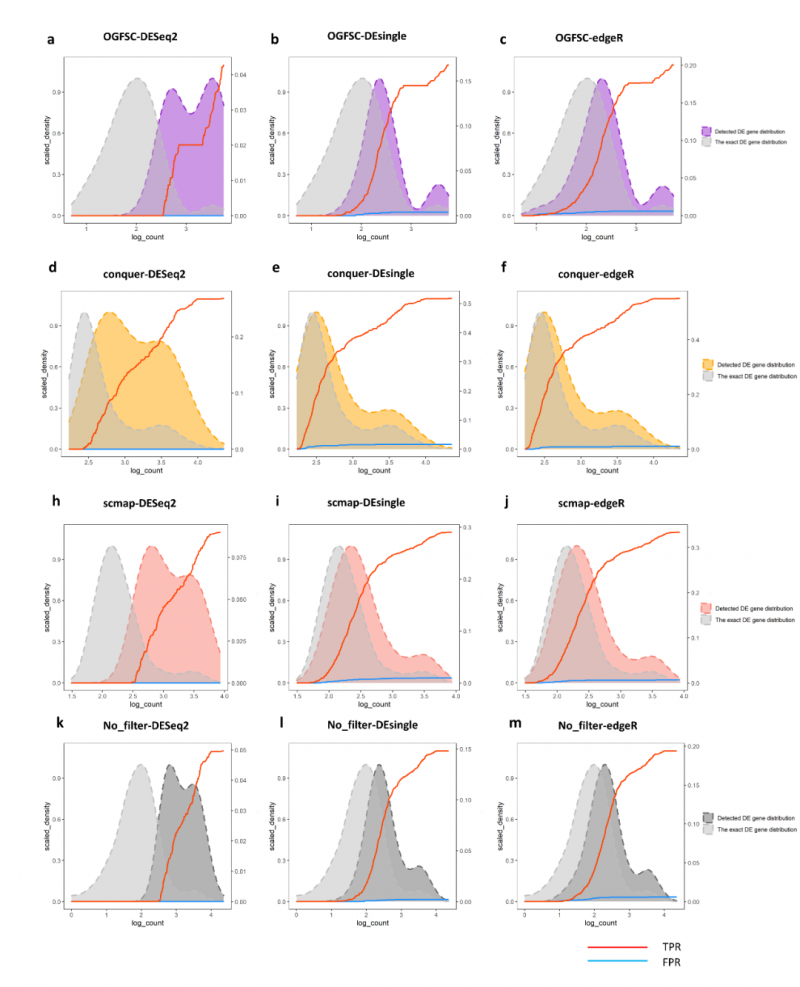
图 2:Investigation of DE gene distribution detected by different combinations of methods, based on simulated-EMTAB8077_heart_kidney.
实际应用中,探索未知领域的 scRNA-seq 样本,其概率分布不同,且缺乏先验知识,无法通过已有的评估标准如 AUROC 去挑选好的分析方法。在本研究中,研究者设计了新的评估参数 CDO 和 AUCC,可以在无需先验知识的创新性研究中,获得针对不同数据的分析策略,更高的真正例率(TPR)和很低的假阳性率(FPR)。
在对小鼠肺癌发育 CD4+ T 细胞分析时,好的分析策略检测到的 DE 基因可以清楚地区分两组细胞,并且随着使用更多基因,细胞聚类的更加清晰。然而,差的分析策略的结果并非如此,未激活的 CD4+ T 细胞仍与活化的 CD4+ T 细胞聚集在一起(图 3)。同时在小鼠心脏和肾脏内皮细胞以及人类肺癌 macrophage m1 m2 数据分析中也得到了类似的结果。
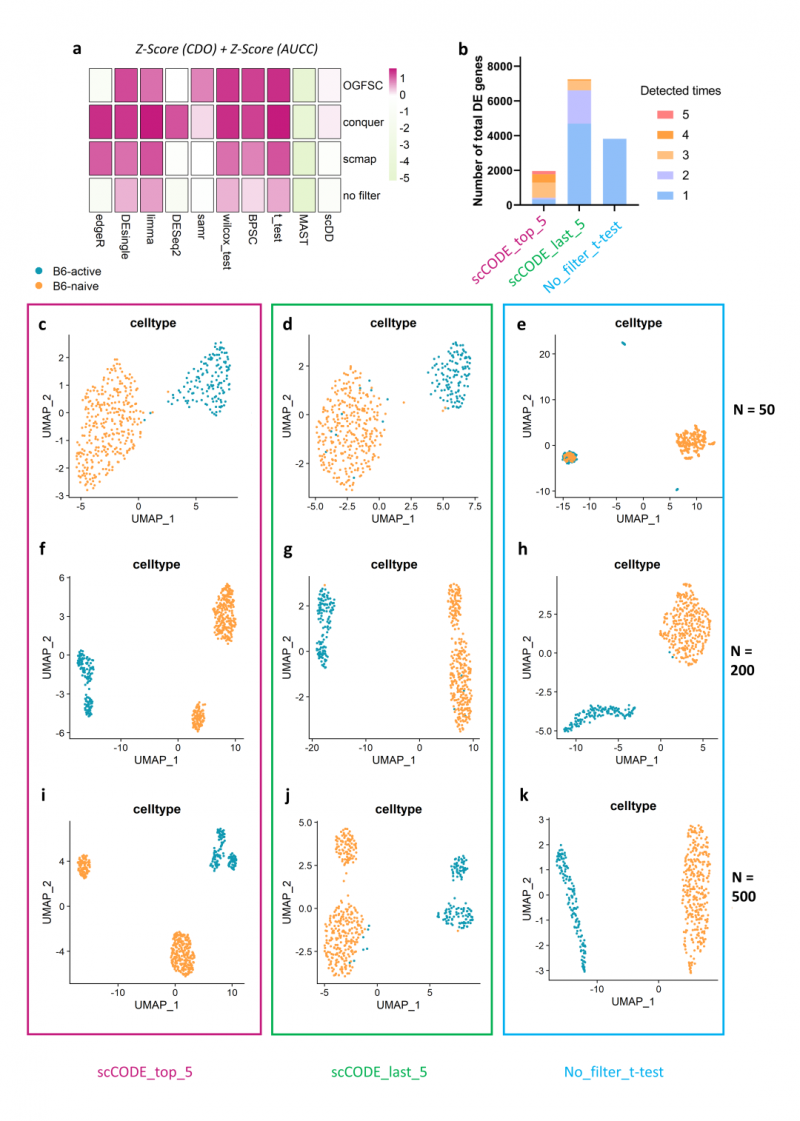
图 3:scCODE validated on real scRNA-seq data comparing activated CD4+ T cells with naïve cells.
该平台已经发布于 GitHub(https://github.com/XZouProjects/scCODE)。目前已收到多个用户反馈良好。论文的通讯作者为复旦大学附属中山医院郝洁研究员,复旦大学附属金山医院邹欣副研究员和辰山植物园陈可副研究员。上海交通大学生命科学技术学院 2019 级硕士邹佳伟为论文第一作者。
该研究得到了国家自然科学基金(82170045,31800253),上海交通大学医学院高水平地方高校创新团队 (SSMU-ZLCX20180502) 上海市绿化和市容管理局科研专项(G222410)等项目支持。
全文链接:https://academic.oup.com/bib/advance-article-abstract/doi/10.1093/bib/bbac180/6590434


