英文题目:A composite single-nucleotide polymorphism prediction signature for extranodal natural killer/T-cell lymphoma
发表期刊:Blood(IF 17.543)
通讯作者:中山大学肿瘤防治中心内科蔡清清教授
DOI:10.1182/blood.2020010637

2021 年 3 月 16 日,中山大学肿瘤防治中心内科蔡清清教授课题组在国际血液学期刊 Blood 上发表了题为:“A composite single-nucleotide polymorphism prediction signature for extranodal natural killer/T-cell lymphoma”的文章。蔡清清教授为通讯作者,中山大学肿瘤防治中心田小朋副研究员、马淑云博士,美国杜克大学 Ken H. Young 教授,新加坡国立癌症中心 Choon Kiat Ong 教授,广东省人民医院刘艳辉教授为共同第一作者。该国际回顾性多中心队列研究首先使用高通量 SNP 芯片(Illumina ASA,由伯豪生物提供服务)鉴定出与生存相关的 36 个 SNP 位点。进一步采用 LASSO 回归模型构建了一个基于 7 个 SNP 的预测模型。创建的 ENKTL 单核苷酸多态性(Signal nucleotide polymorphism, SNP) 预后评价系统修正了肿瘤异质性及活检取材部位的偏差,作为有效工具个体化预测 ENKTL 患者预后,可用于指导早期高危患者接受联合放化疗。该评价系统可为不同预后风险的 ENKTL 患者指导精准治疗决策。
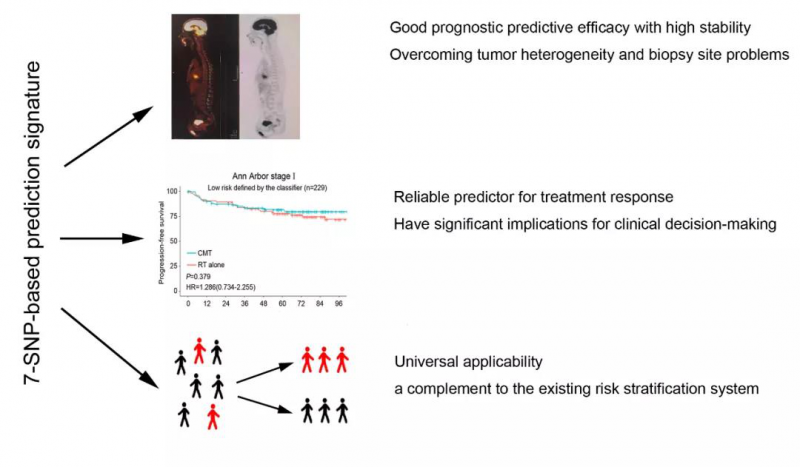
摘要:当前基于临床病理变量的预后评分系统不足以预测接受非蒽环类药物治疗的结外鼻型 NK/ T 细胞淋巴瘤(ENKTL)患者的生存和治疗反应。这项国际回顾性多中心队列研究旨在构建基于单核苷酸多态性(SNP)的分类器,以提高预测准确性并指导临床决策。分析了来自国际多中心的 722 名 ENKTL 患者的数据,使用 LASSO 回归在训练集(n = 336)中构建了基于 7 -SNP 的分类器,并在内部测试集(n=144)和两个外部验证集(n=142; n=100)中进一步进行了验证。基于 7 -SNP 的分类器在训练集和三个验证集中显示出良好的预后预测功效。由分类器计算的高风险评分和低风险评分的患者表现出显着不同的无进展生存期(PFS)和总体生存期(OS)(所有 p <0.001)。通过多变量分析进一步证明了基于 7 -SNP 的分类器是一个独立的预后因素,其预测准确性明显优于临床病理风险变量。基于 7 -SNP 的分类器的应用不受样品类型及肿瘤异质性的影响。值得注意的是,与单纯放疗相比,在高风险的 Ann Anbor I 期患者中,化学疗法联合放疗的显著改善了 PFS 和 OS,而在低危患者中,两种治疗方式之间没有统计学差异。由分类器和临床病理变量结合的 nomogram 评分系统明显提高了预测的准确性。基于 7 -SNP 的分类器是对 ENKTL 中现有风险分层系统的补充,这可能会对 ENKTL 患者的临床决策产生重大影响。
更多伯豪生物人工服务:


